
9月15日,真迈生物与序祯达生物、UST共同签署协议,正式达成战略合作。根据协议,序祯达生物将采用UST的TELL-Seq关联长读长二代测序技术,并通过真迈生物的GenoLab M高通量基因测序平台,推出经济可及的长读长测序解决方案,为广大合作伙伴提供高品质的检测分析服务。作为此次战略合作的第一个重量级成果,三方联合推出的「细菌基因组完成图-NGS跃级版」今日发布。
它的详情,来一睹为快↓↓↓
细菌基因组完成图-NGS跃级版
组装细菌完整基因组是现代微生物研究的重要手段。利用细菌基因组完成图可以更好地研究重要基因功能、鉴定未知功能基因以及比较物种间的进化关系,这在传染性疾病的研究、预防与治疗,环境微生物研究及疫苗和抗生物等的研究开发中都发挥着重要作用。
序祯达生物采用UST的TELL-Seq关联长读长二代测序技术,结合真迈生物的GenoLab M高通量基因测序平台,打造出一款通过二代测序平台即能接近三代组装效果的全新产品——细菌基因组完成图-NGS跃级版。
丨长读长二代测序技术让细菌基因组完成图绘制触手可及
细菌基因组完成图-NGS跃级版采用了关联长读长二代测序技术——TELL-Seq,该技术通过转座酶对来自同一条长DNA的小片段进行特定标记,利用标记对短读长序列进行拼接,让二代测序的短读长数据产生超长读长测序效果,等价读长达20 kb-200 kb。经过创新开发的生信算法,组装的平均效果为contig(>10kb):1-10个;N50:2-5Mb(与基因组大小关联)。
该技术不仅可用于微生物de novo测序(菌种鉴定),还可应用于基因组单倍体分型(Phasing)、基因组结构变异、新物种全基因组de novo组装等多个方向。

丨细菌基因组完成图-NGS跃级版,揭示细菌基因组完整面貌
细菌基因组完成图-NGS跃级版通过基于TELL-Seq的二代测序技术以真迈生物的GenoLab M为载体,对细菌基因组进行测序,实现高性价比的基因组完成图组装,获得高准确度的基因组信息。

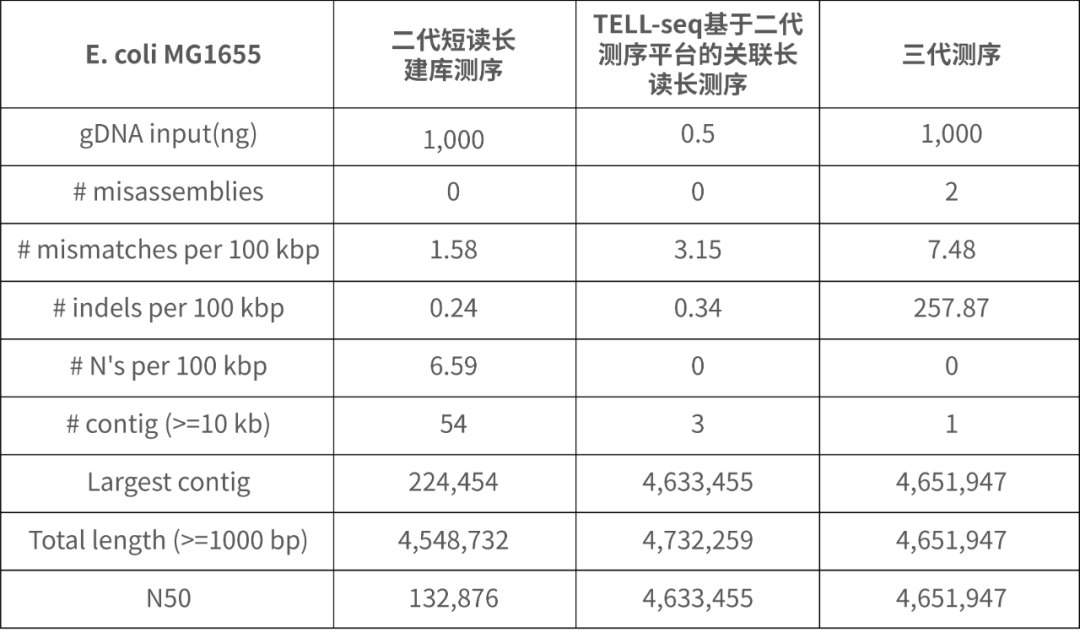
丨卓越效果,眼见为实
TELL-Seq技术的测序数据可以组装出接近完美的细菌基因组
样本要求及检测周期:
送样要求:≥50ng,片段长度>20kb,未经反复冻融,无降解,无污染
运输要求:-80℃保存,干冰运输
检测周期:最短3周(收到样本至交付组装结果)
交付结果:DNA质检报告、测序下机原始数据、组装结果
关于GenoLab M
GenoLab M是一款精准、高效、灵活、开放的桌面型高通量基因测序平台,其采用基于芯片扩增的表面荧光测序技术SURFSeq对碱基的荧光信号进行识别,实现边合成边测序。GenoLab M可以兼容目前市面上主流的NGS文库制备产品和数据分析流程。
相较于目前市场上的同类竞品,GenoLab M在拥有双芯片平台的基础上,还支持'滚动上机'模式,“滚动上机”是指GenoLab M在运行单侧芯片平台测序时,用户可根据实际实验需求随时启动空闲芯片平台的测试工作,“双芯片平台+滚动上机模式”极大地提高了GenoLab M的检测灵活性,为用户带来了优秀的使用体验。

参考文献:
1. Chen Z, Pham L, Wu TC, et al. Ultra-low input single tube linked-read library method enables short-read second-generation sequencing systems to generate highly accurate and economical long-range sequencing information routinely. Genome Res. 2020;30(6):898-909. doi: 10.1101/gr.260380.119.
2. Arun Sethuraman, Rosalina Stancheva, Ciara Sanders, et al. Genome of a novel Sediminibacterium discovered in association with two species of freshwater cyanobacteria from streams in Southern California, G3 Genes|Genomes|Genetics, Volume 12, Issue 7, July 2022, jkac123, https://doi.org/10.1093/g3journal/jkac123
3. Kumar, A., Im, K., Banjevic, M. et al. Whole-genome risk prediction of common diseases in human preimplantation embryos. Nat Med 28, 513–516 (2022). https://doi.org/10.1038/s41591-022-01735-0
4. Faith M Anderson, Noelle Visser, Kevin Amses, et al. Human commensal Candida albicans strains demonstrate substantial within-host diversity and retained pathogenic potential. bioRxiv 2022.09.09.507247; https://doi.org/10.1101/2022.09.09.507247